目录
最后一次练习,对应课程结尾的对数线性模型框架;于是又拿下一门课。在这次练习中,我们将使用感知机算法训练一个GLM应用到命名实体识别上。对输入实例![]() ,GLM使用如下三个组件完成解码:
,GLM使用如下三个组件完成解码:
-
一个函数生成所有可能的结果
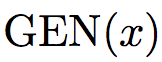
-
一个全局特征函数
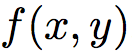
-
一个参数向量
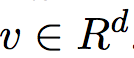
解码问题定义为:
![]()
课上讲过如果全局特征可拆分为局部特征的话,即使![]() 很大,解码依然高效。具体到标注问题,我们将整个标注结果拆分为一个历史片段序列
很大,解码依然高效。具体到标注问题,我们将整个标注结果拆分为一个历史片段序列![]() ,然后将
,然后将![]() 分解为定义在每个历史片段和标签上的局部特征函数之和,比如
分解为定义在每个历史片段和标签上的局部特征函数之和,比如![]() 。这里我们使用trigram模型,所以定义历史片段为:
。这里我们使用trigram模型,所以定义历史片段为:
![]()
于是引入局部特征的解码问题定义为:
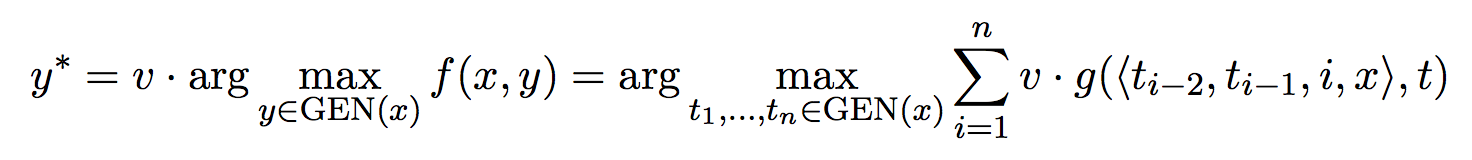
在这次练习中,我们将实验不同的特征函数,并且实现感知机算法。
数据
数据和评测与第一次练习HMM一样(我恰好跳过了那次练习)。
数据是来自BioCreAtIvE II的基因名称识别,由训练集和开发集组成。经典的IO标签,示例如下:
Comparison O with O alkaline I-GENE phosphatases I-GENE and O 5 I-GENE - I-GENE nucleotidase I-GENE Pharmacologic O aspects O of O neonatal O hyperbilirubinemia O .O ……
对应的读取代码
def read_sentence_tags(doc_fpath):
"""where the document separates each token (including newlines) into its own line"""
sentences = []
tag_lines = []
with open(doc_fpath, 'r') as pile:
sentence = []
tags = []
for line in pile:
tup = line.strip().split(' ')
if len(tup) == 1: # end of sentence
sentences.append(sentence) if sentence else None # ignore newline only sentences
tag_lines.append(tags) if tags else None
sentence = []
tags = []
else:
word, tag = tup
sentence.append(word)
tags.append(tag)
return sentences, tag_lines
历史信息
在即将看到的第一题中,我们先假设参数向量是现成并固定的,方便新手上路。目标仅要求利用该向量解决解码问题。考虑如下句子
Characteristics of lipase activity .
它的历史信息是什么样的呢?对i=3这个时刻,可能的历史信息如下:
⟨I-GENE,I-GENE,x,3⟩ ⟨O,I-GENE,x,3⟩ ⟨I-GENE,O,x,3⟩ ⟨O,O,x,3⟩
因为标注集大小为2,那么前两个标签的组合就有2*2=4种了。
然后分别拼接两种标签即为局部特征函数的输入了。
特征向量
GLM的艺术在于挑选好的特征,一个典型的特征类似如下:
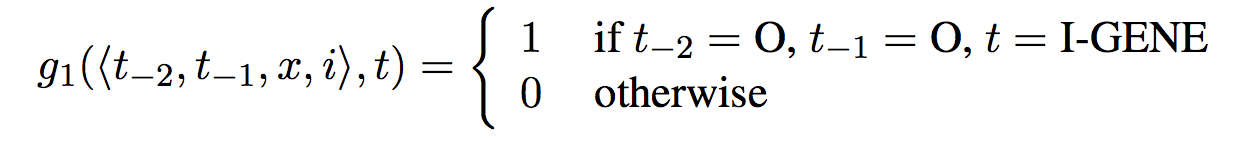
尖括号内就是历史信息,这里是前两个标签。x其实是整个句子,在实现上可以省略掉。取个友好的名字,就叫标签上的三元文法吧:![]() 。
。
一旦有了特征集,就可以对一个特定的历史信息/标签对求出所有特征函数的输出,该输出组成了一个01向量![]() ,与参数向量做内积得到score
,与参数向量做内积得到score![]() 。
。
解码
为了找出一个句子的最佳全局标签序列,需要解决如下解码问题:
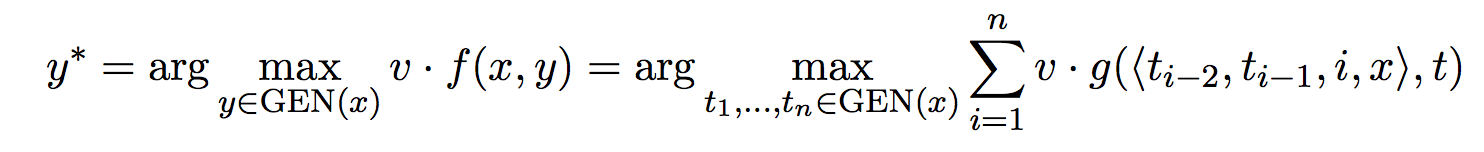
这里为了教学方便,仅使用了一种特征,即标签上的trigram。
定义为![]() 标注集全集,定义
标注集全集,定义
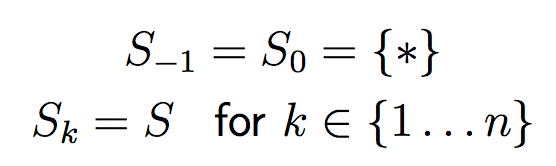
GLM的解码算法依然是viterbi:

该变种与HMM-viterbi唯一的不同是score函数的不同,HMM中是转移矩阵+发射矩阵,此处是特征向量与参数向量的内积![]() 。
。
问题1
请利用已训练好的模型实现trigram解码器。该模型利用到的特征非常非常简单,只有两种:
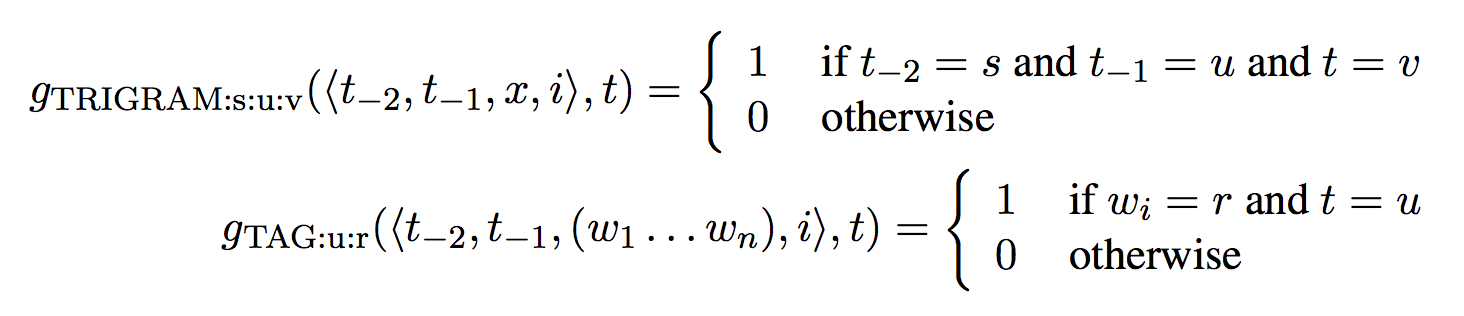
即1、标签上的trigram;2、当前词语。
比如上面那个例子,在i=3,y=I-GENE时的特征向量为
![]()
其他维度都是0。
已训练的模型格式如下:
TAG:lipase:I-GENE 0.23 TRIGRAM:O:O:I-GENE 0.45
加载模型
按行读取feature-weight pair,空格分割:
def read_tag_model(file_path):
model = {}
with open(file_path, 'r') as pile:
for line in pile:
feature, weight = line.strip().split(' ')
model[feature] = float(weight)
return model
特征抽取
由于模型中只有两种特征,所以只需实现这两种特征即可:
def trigram_feature(a, b, c):
"""
trigram feature generator
:param a: first tag
:param b: second tag
:param c: third tag
:return: string representation
"""
return 'TRIGRAM:{}:{}:{}'.format(a, b, c)
def tag_feature(word, tag):
"""
tag feature generator
:param word:
:param tag:
:return:
"""
return 'TAG:{}:{}'.format(word, tag)
特征抽取时得到的是两者的集合,代表着一个01向量v:
def simple_feature(w, u, v, word): """ simple feature extractor :param w: -2 tag :param u: -1 tag :param v: current tag :param word: current word :return: """ feature = [trigram_feature(w, u, v), tag_feature(word, v)] return feature
内积
对特征向量g,与参数向量做内积![]() :
:
def vg(w, u, v, word, dict_tag_model): """ v dot g for current state :param w: -2 tag :param u: -1 tag :param v: current tag :param word: current word :param dict_tag_model: tag model :return: """ v = simple_feature(w, u, v, word) ## dot product on weights and if it exists vg = sum([dict_tag_model.get(k, 0) for k in v]) return vg
viterbi解码
写了无数遍了……
对照伪码:

不难理解以下代码:
def viterbi(sentence, dict_tag_model):
"""calculate y^* = \arg\max_{t_1,\dots,t_n \in GEN(x)} f(x,y) \cdot v. x is the sentence history"""
n = len(sentence)
S = ['I-GENE', 'O']
pie, bp = {(0, '*', '*'): 0}, {}
def S_(k):
return ['*'] if k <= 0 else S
for k in xrange(1, n + 1):
word = sentence[k - 1] # idx-0
for u, s in product(S_(k - 1), S_(k)):
## \max_{w \in S_{k-2}}
max_val, max_tag = -float('inf'), None
for t in S_(k - 2):
vg_ = vg(t, u, s, word, dict_tag_model)
pie_ = pie[(k - 1, t, u)]
## previous most likely sentence so far, then probability of this trigram
pkus = pie_ + vg_
if pkus > max_val:
max_val, max_tag = pkus, t
idx = (k, u, s)
pie[idx], bp[idx] = max_val, max_tag
## calculate for the end of sentence
max_val, max_tag = -float('inf'), None
for u, s in product(S_(n - 1), S_(n)):
pkus = pie[(n, u, s)] + vg(u, s, 'STOP', word, dict_tag_model)
if pkus > max_val:
max_val, max_tag = pkus, (u, s)
## go back in the chain ending with y_{n-1} and y_n
t = [None] * n
t[n - 1] = max_tag[1] # y_n
t[n - 1 - 1] = max_tag[0] # y_{n-1}
for k in xrange(n - 2, 1 - 1, -1):
t[k - 1] = bp[(k + 2, t[(k + 1) - 1], t[(k + 2) - 1])]
return t
调用方法
sentences, tag_lines = read_sentence_tags(gene_fpath)
dict_tag_model = read_tag_model(args.model)
for sentence in sentences:
tags = viterbi(sentence, dict_tag_model)
for word, tag in zip(sentence, tags):
sys.stdout.write('{} {}\n'.format(word, tag))
sys.stdout.write('\n')
评测
执行
#!/usr/bin/env bash python glm.py p1 > gene_dev.p1.out python eval_gene_tagger.py gene.key gene_dev.p1.out
得到
Found 1337 GENEs. Expected 642 GENEs; Correct: 280. precision recall F1-Score GENE: 0.209424 0.436137 0.282971
为什么这么差呢,因为提供的这个模型使用的特征与传统的HMM一模一样,没有利用到全局特征。
感知机训练算法
第一题中的模型是预先提供的,如何自己训练呢?注意不同于HMM,我们不能简单地读取训练数据做些简单的统计就能得到模型参数。我们将反复地执行解码,根据结果调整参数。
举个例子,训练语料中有个句子:
Characteristics O of O lipase I-GENE activity O .O
在单词lipase下标(时刻)i=3的时候,有历史/标签pair:
(⟨O,O,x,3⟩,I-GENE)
如果某个特征函数对
(⟨O,O,x,3⟩,O)
输出了1,那么该特征函数不可信,它的weight应该减少。
形式化地讲,训练算法如下:
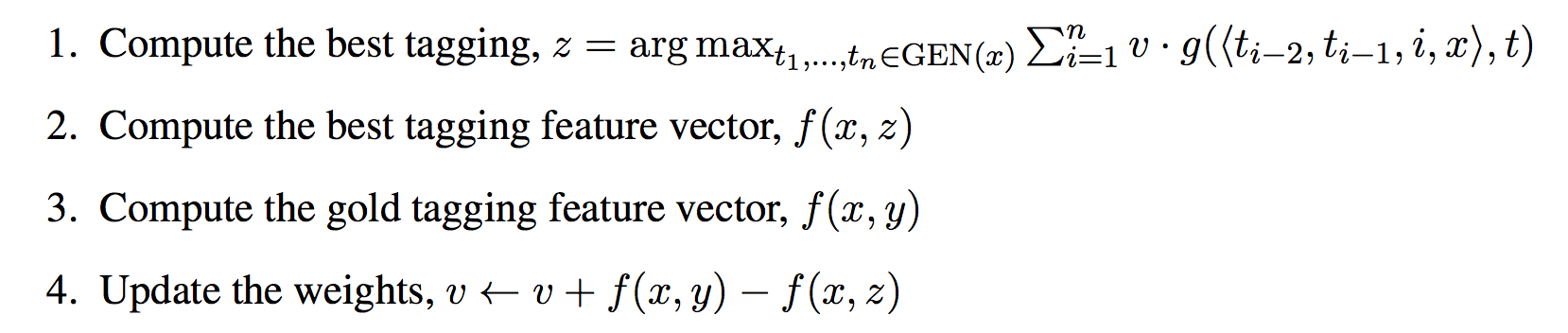
问题2
问题1中的模型弱点在于特征,它无法处理低频词。我们可以像HMM一样引入_RARE_标记,但在GLM中可以通过引入更多特征来捕捉词语的某个特性。
一个不错的例子是利用单词的后缀,虽然模型从没见过某个词,但它很可能见过相同的后缀(英语的词根词缀),根据后缀大概就能猜出单词的意思。
该特征函数定义如下:
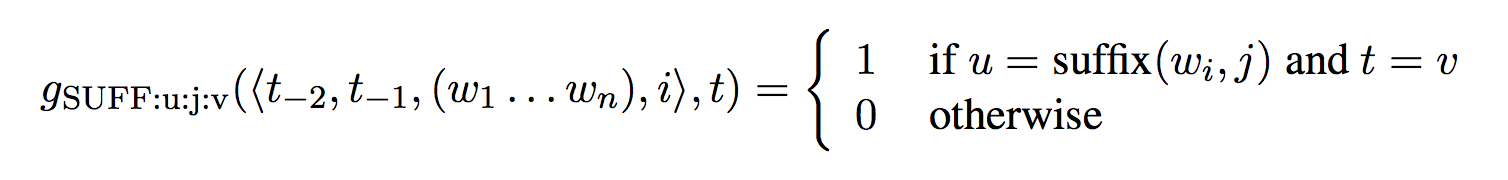
其中,![]() 表示单词w的长j的后缀。
表示单词w的长j的后缀。
后缀特征
用Python实现如下:
def suff_feature(word, tag):
"""
suffix feature function
:param word:
:param tag:
:return:
"""
suff_keys = []
for idx in range(1, 3 + 1):
suff = word[-idx:]
if len(suff) != idx: # ie. not enough letters remaining
continue
suff_keys.append('SUFF:{}:{}:{}'.format(suff, idx, tag))
return suff_keys
拓展特征函数集合
将simple_feature替换为more_feature:
def more_feature(w, u, v, word): """ simple feature extractor :param w: -2 tag :param u: -1 tag :param v: current tag :param word: current word :return: """ feature = simple_feature(w, u, v, word) feature += suff_feature(word, v) return feature
感知机算法
同样写过了几遍……
对照伪码
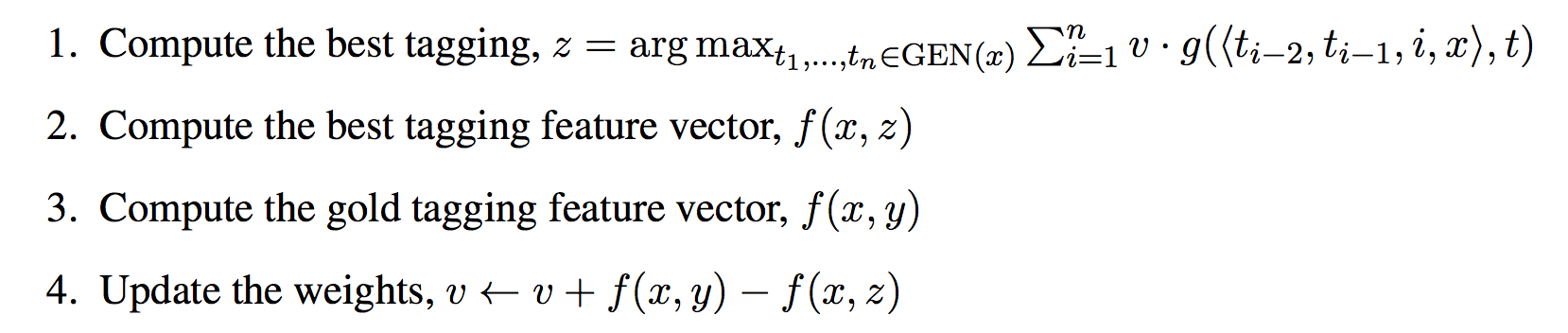
实现如下:
def perceptron(sentences, tag_lines, n_iter): model = defaultdict(int) # v def feature_vector(tags, sentence): v = defaultdict(int) tags = ['*', '*'] + tags + ['STOP'] for word, w, u, s in zip(sentence, tags, tags[1:], tags[2:]): for f in most_feature(w, u, s, word): v[f] += 1 return dict(v) for _ in xrange(n_iter): for sentence, gold_tags in zip(sentences, tag_lines): best_tags = viterbi(sentence, model) # best tag sequence (GEN) under the current model gold_features = feature_vector(gold_tags, sentence) best_features = feature_vector(best_tags, sentence) if gold_features != best_features: ## update weight vector v for key, val in gold_features.iteritems(): model[key] += val for key, val in best_features.iteritems(): model[key] -= val return model
调用方法
sentences, tag_lines = read_sentence_tags(gene_fpath)
dict_tag_model = perceptron(sentences, tag_lines, 6)
for sentence in sentences:
tags = viterbi(sentence, dict_tag_model)
for word, tag in zip(sentence, tags):
sys.stdout.write('{} {}\n'.format(word, tag))
sys.stdout.write('\n')
评测
Found 775 GENEs. Expected 642 GENEs; Correct: 390. precision recall F1-Score GENE: 0.503226 0.607477 0.550459
疗效显著。
问题3
GLM的优势在于能够利用丰富的全局特征,因为其历史是建立在整个句子之上的。请自己定义一些特征,观察对准确率的影响。注意有些特征反而会降低准确率;特征工程需要特定的领域知识,有时候挺难的。
观察到一些基因含有特殊的标点符号:
5 I-GENE - I-GENE nucleotidase I-GENE
所以可以将标点符号作为一种特征。
标点符号特征
def gene_punc_feature(word, w, u, v):
if word in '-/()\'.':
return 'GENE_PUNC:{}:{}:{}:{}'.format(word, w, u, v)
将more_feature替换为:
def most_feature(w, u, v, word): """ simple feature extractor :param w: -2 tag :param u: -1 tag :param v: current tag :param word: current word :return: """ feature = simple_feature(w, u, v, word) feature += suff_feature(word, v) punc_feature = gene_punc_feature(word, w, u, v) if punc_feature: feature.append(gene_punc_feature(word, w, u, v)) return feature
即可。
评测
Found 571 GENEs. Expected 642 GENEs; Correct: 366. precision recall F1-Score GENE: 0.640981 0.570093 0.603462
果然有效,说明追求分数的话,多加特征吧。
Reference
https://github.com/hankcs/Coursera_NLP_MC
https://github.com/leungwk/nlangp
 码农场
码农场
有公开课的视频吗?coursera上现在看不到了, 网上的分享链接也都失效了
我是从网盘搜索引擎里面找到的